
2025年3月19日,我院天然药物及仿生药物全国重点实验室乔雪研究员团队与英国John Innes Centre的Anne Osbourn教授团队合作,在国际知名期刊Proc. Natl. Acad. Sci. U. S. A.发表了题为“Comprehensive mutant chemotyping reveals embedding of a lineage-specific biosynthetic gene cluster in wider plant metabolism”的研究论文,揭示了植物广泛代谢网络对谱系特异性生物合成基因簇的整合作用。

植物合成多种多样的次生代谢物,这些代谢物具有重要的生理意义和药理活性。越来越多的研究表明,编码这些代谢途径的基因在植物基因组中并非分散分布,而是成簇存在。在植物中,生物合成基因簇编码的代谢途径通常是独立的线性通路,不参与更广泛的代谢网络。相比之下,类胡萝卜素和花青素等广泛分布的保守代谢物,其生物合成基因则呈非簇状分布。对于这些新近进化形成的生物合成基因簇如何与植物基础代谢网络相互作用,目前知之甚少。
针对以上科学问题,我们利用Osbourn教授课题组前期鉴定的一个生物合成基因簇作为研究对象,它由12个基因组成,负责合成燕麦的抗菌防御化合物——三萜皂苷avenacin A-1。本研究通过筛选100余个avenacin缺陷型突变体,结合突变体化学分型、生化分析和基因组重测序技术,鉴定了两个非簇状分布的基因Sad4和Pal2。
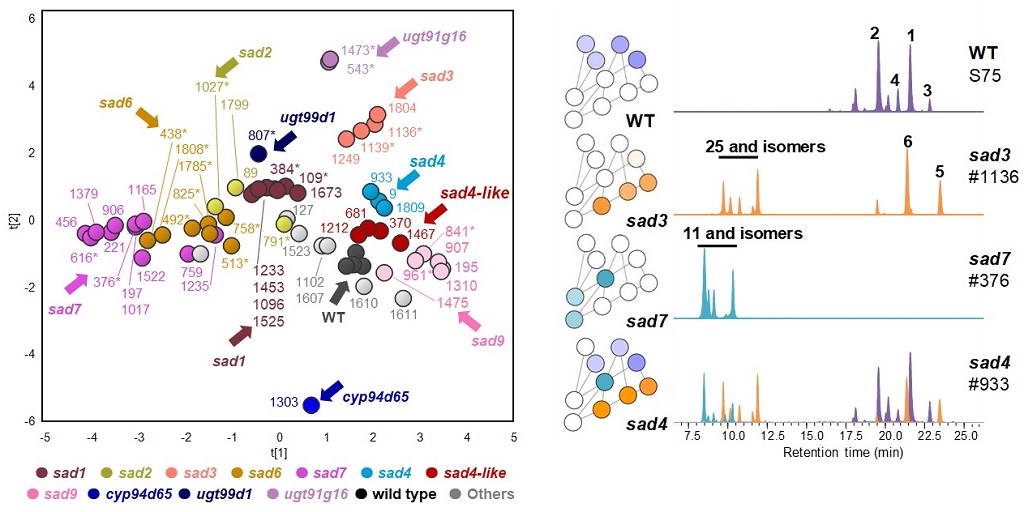
图1. 突变体化学分型鉴定Sad4和Sad4-like(即Pal2)表型
通过体内、体外生化实验表明,SAD4是苯丙烷和色氨酸途径的下游糖基转移酶,可催化来自苯丙烷和色氨酸途径的底物形成葡萄糖酯键,是UGT84家族中底物谱最广的成员之一。PAL2是苯丙烷途径中的第一个关键酶,其下游产物可生成多种酚类骨架结构。SAD4和PAL2在燕麦体内负责合成avenacin糖基化和酰基化所需的供体分子。
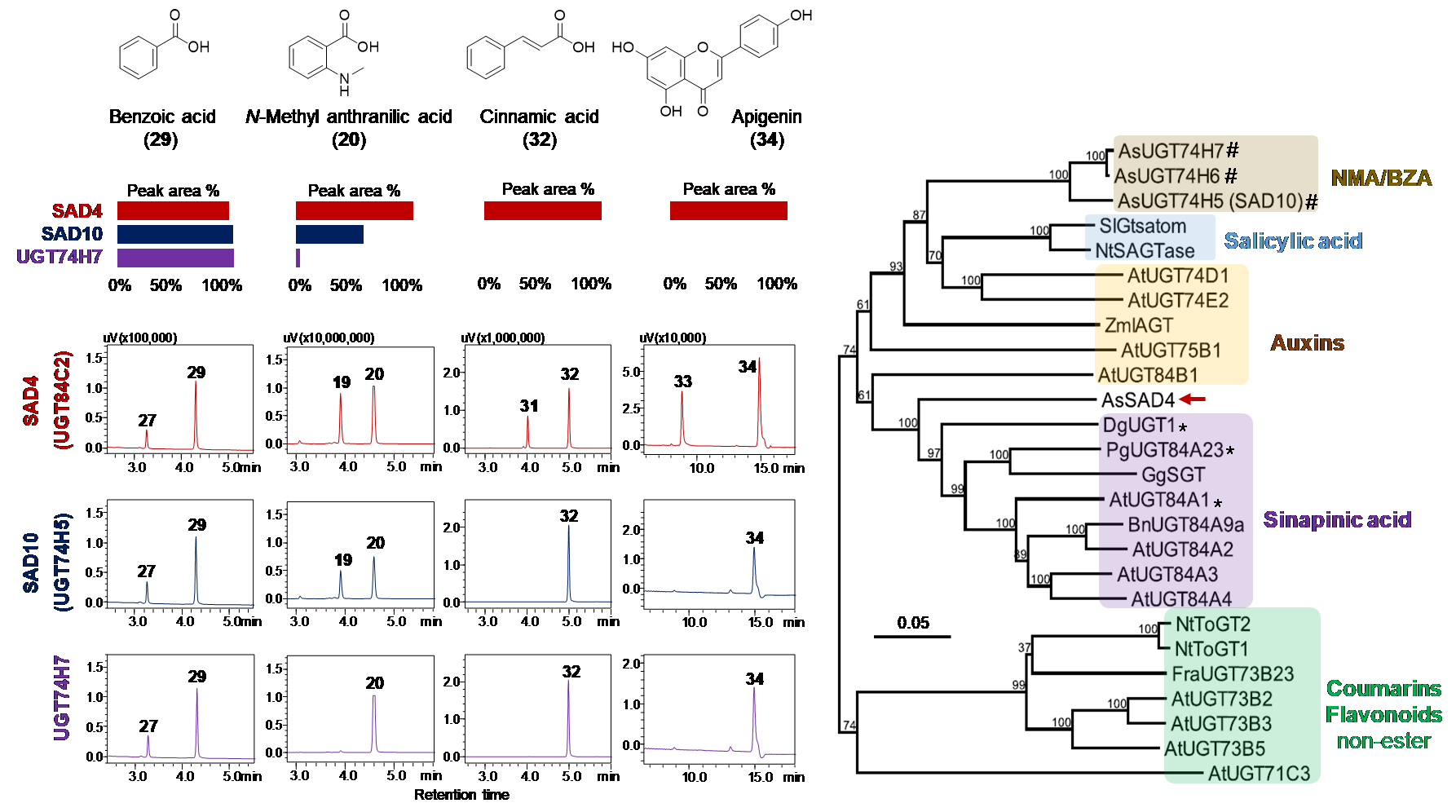
图2. SAD4的催化功能及序列特异性
与avenacin生物合成基因簇中的基因相比,SAD4和PAL2在燕麦中表现出更广泛的表达谱,并较其同工酶参与更多样的生化反应。这两个酶是从苯丙烷和色氨酸途径中招募而来,属于基础代谢网络的一部分。相应的基因不成簇分布,与avenacin生物合成基因簇的表达谱不同,且对植物的木质素合成等初生代谢没有显著影响。由于这些酶不属于avenacin核心合成途径,而是为avenacin的酰基化和糖基化提供前体,我们将其定义为簇辅助酶(CAEs)。这些簇辅助酶的发现,为理解植物基础代谢与新近进化的谱系特异性生物合成基因簇之间的相互作用机制提供了新视角。
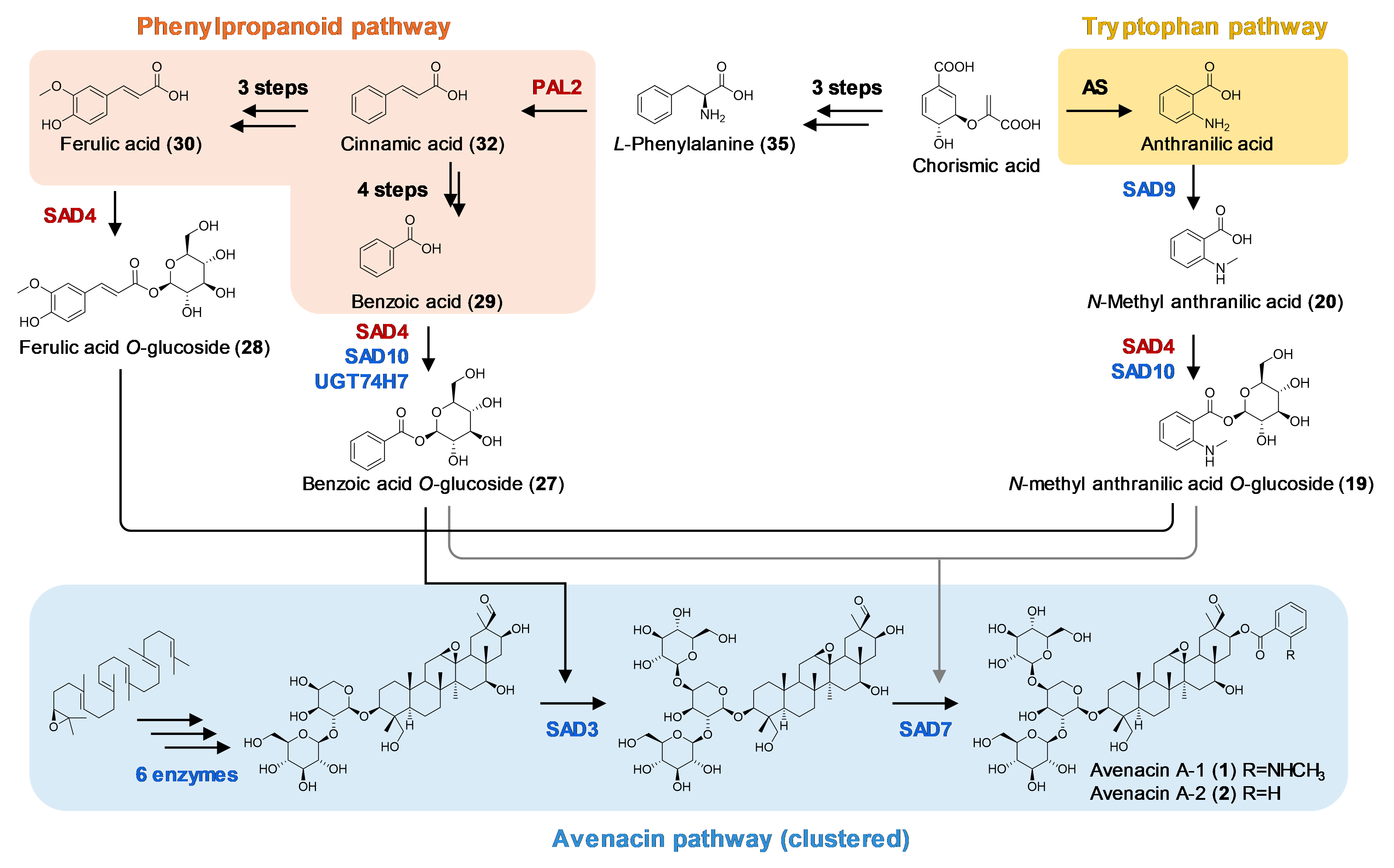
图3. 簇辅助酶SAD4和PAL2在三萜皂苷avenacin A生物合成途径中的功能
乔雪研究员为本论文的第一作者兼通讯作者,英国John Innes Centre的Anne Osbourn教授为本文的最后通讯作者。药学院2021级直博生张嘉贺参与了本论文工作。药学院叶敏教授为本论文工作的顺利开展提供了重要帮助。该工作得到了国家自然科学基金项目的资助。
论文链接:https://www.pnas.org/doi/10.1073/pnas.2417588122
【乔雪研究员简介】

乔雪,北京大学药学院研究员,博雅特聘教授,博士生导师。2012年毕业于北京大学药学院,获博士学位,后留校任教,2016-2018年在英国John Innes Centre做访问学者。主要研究领域为中药药效成分及其生物合成,对黄芪皂苷生物合成途径开展了系列研究。在Nat Commun、PNAS、J Am Chem Soc、Angew Chem Int Ed等期刊发表SCI论文90余篇,获批专利10余项。入选国家基金委杰青、优青、北京市杰青、中国科协青年人才托举工程等人才计划。
天然药物及仿生药物全国重点实验室